Capítulo 2 Modelo lineal múltiple
Grasa corporal
Retomemos la base de datos de grasa corporal. Aparte de la circunferencia del abdomen, el % de grasa corporal de los hombres puede estar explicado con otros factores. Por ejemplo, la edad, el peso, la estatura, entre otros. En la base de datos vamos a considerar la circunferencia del abdomen (abdom), la edad (age) y el índice de masa corporal (adipos) como covariables para explicar el % de grasa corporal (brozek).
La Figura 2.1 muestra la relación entre las variables de estudio. Aquí podemos observar que el % de grasa corporal se relaciona de forma lineal positiva fuerte con la circunferencia del abdomen (correlación de 0.81) y el peso (correlación igual a 0.61). Mientras que la relación con la edad no es debil (correlación de 0.29).
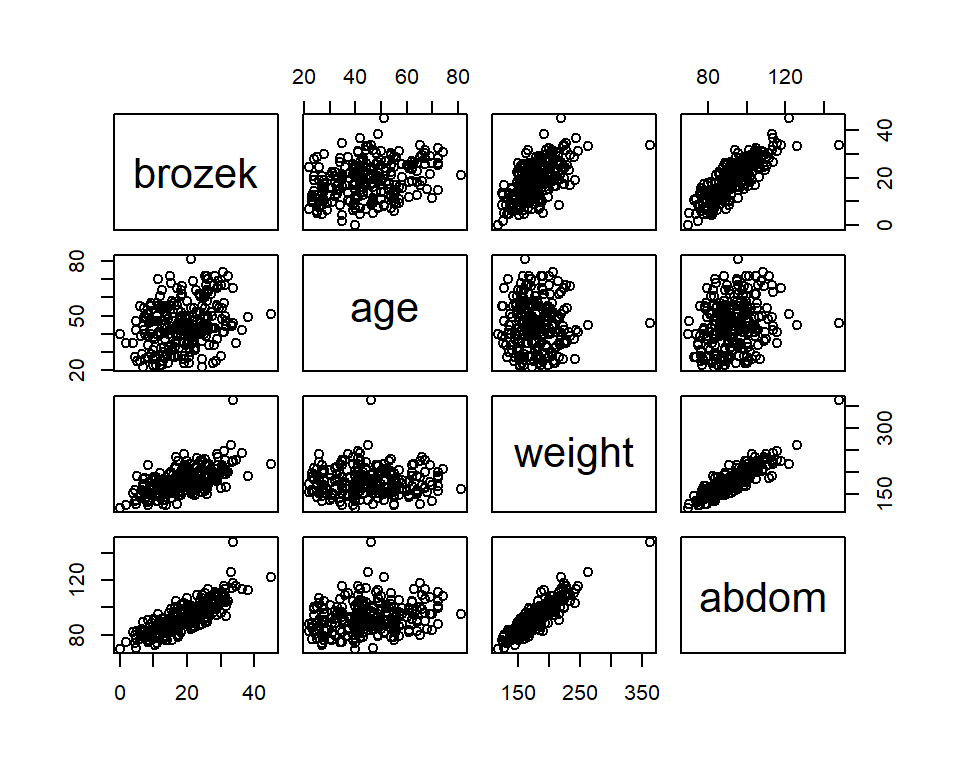
Figure 2.1: Gráfico de dispersion del % de grasa corporal en función de la edad(age, en años), peso(weight, en kg) y la circunferencia dle abdomen(abdom, en cm).
## brozek age weight abdom
## brozek 1.0000000 0.28917352 0.61315611 0.8137062
## age 0.2891735 1.00000000 -0.01274609 0.2304094
## weight 0.6131561 -0.01274609 1.00000000 0.8879949
## abdom 0.8137062 0.23040942 0.88799494 1.0000000Por lo tanto, junto con la circunferencia del abdomen, ahora vamos a incluir peso (en kg, weigth) y la edad (en años, age) como covariable. Por lo tanto, el modelo propuesto es:
\[
\mbox{brozek}_{i} = \beta_{0} + \beta_{1}\mbox{abdom}_{i} + \beta_{2}\mbox{weigth}_{i}+\beta_{3}\mbox{age}_{i} + \varepsilon_{i},
\]
donde \(\varepsilon_{i}\sim N(0,\sigma^{2})\), y \(cov(\varepsilon_{j},\varepsilon_{k})=0\) para todo \(j\neq k\).
2.1 Modelo lineal múltiple
En general, se puede relacionar la variable respuesta (\(y\)), con \(p-1\) covariables (o variables predictoras). El modelo lineal múltiple se expresa de la siguiente forma:
\[\begin{equation} \begin{split} y_{i} =& \beta_{0} + \beta_{1}x_{i1} + \beta_{2}x_{i2} + \ldots + \beta_{p-1} x_{i,p-1} + \varepsilon_{i} \\ =& \boldsymbol x_{i}'\boldsymbol \beta+ \varepsilon_{i}, \qquad i=1,\ldots,n, \\ \end{split} \tag{2.1} \end{equation}\]
donde \(\boldsymbol x_{i} = (1,x_{i1},x_{i2},\ldots,x_{i,p-1})'\) es el vector de dimensión \(p\) de covariables del individuo \(i\) y \(\boldsymbol \beta= (\beta_{0},\beta_{1},\ldots,\beta_{p-1})'\) es el vector de dimensión \(p\) de coeficientes de regresión.
Los supuestos del modelo son los mismos que se plantearon en el capítulo anterior. Estos es: \(\varepsilon_{i} \sim N(0,\sigma^{2})\) y \(cov(\varepsilon_{i},\varepsilon_{j})=0\) para \(i \neq j\).
Dado que \(E(\varepsilon_{i})=0\), el valor esperado de \(Y\) es:
\[\begin{equation} E(Y| \boldsymbol x_{i}) = \beta_{0} + \beta_{1}x_{i1} + \beta_{2}x_{i2} + \ldots + \beta_{p-1} x_{i,p-1} = \boldsymbol x_{i}'\boldsymbol \beta. \tag{2.2} \end{equation}\]
El intercepto \(\beta_{0}\) es el valor esperado de \(Y\) cuando \(x_{i}=(1,0,0,\ldots,0)'\), es decir cuando todos las covariables toman el valor \(0\).
El parámetro de pendiente \(\beta_{j}\) indica el cambio en el valor esperado de \(Y\) debido a un aumento unitario en la covariable \(x_{j}\) cuando todas las demás variables predictoras se mantienen constantes. Sean \(x_{i,j} = (1,x_{i1},\ldots,x_{ij},\ldots,x_{i,p-1})\) y \(x_{i,j+1} = (1,x_{i1},\ldots,x_{ij}+1,\ldots,x_{i,p-1})\). A partir de (2.2), tenemos:
\[ E(Y|x_{i,j}) = \beta_{0} + \beta_{1}x_{i1} + \ldots + \beta_{j}x_{ij} + \ldots + \beta_{p-1} x_{i,p-1}, \]
y
\[ E(Y|x_{i,j+1}) = \beta_{0} + \beta_{1}x_{i1} + \ldots + \beta_{j}(x_{ij}+1) + \ldots + \beta_{p-1} x_{i,p-1}. \]
De aquí tenemos que: \[ E(Y|x_{i,j+1}) - E(Y|x_{i,j}) = \beta_{j}. \] Es conveniente escribir el modelo de regresión múltiple (2.1) de forma matricial: \[ \boldsymbol y= \boldsymbol X\boldsymbol \beta+ \boldsymbol \varepsilon, \] donde: \[\begin{gather} \begin{aligned} \boldsymbol y= \begin{pmatrix} y_{1} \\ y_{2} \\ \vdots \\ y_{n} \end{pmatrix}, & \boldsymbol X= \begin{pmatrix} 1 & x_{11} & x_{12} & \ldots & x_{1,p-1} \\ 1 & x_{21} & x_{22} & \ldots & x_{2,p-1} \\ \vdots & \vdots & \vdots & \ddots & \vdots \\ 1 & x_{n1} & x_{n2} & \ldots & x_{n,p-1} \end{pmatrix}, \boldsymbol \beta= \begin{pmatrix} \beta_{0} \\ \beta_{1} \\ \beta_{2} \\ \vdots \\ \beta_{p-1} \end{pmatrix}, & \boldsymbol \varepsilon= \begin{pmatrix} \varepsilon_{1} \\ \varepsilon_{2} \\ \vdots \\ \varepsilon_{n} \end{pmatrix}. \end{aligned} \nonumber \end{gather}\] Además, los supuestos sobre los errores se pueden expresar como \(\boldsymbol \varepsilon\sim N(\boldsymbol 0, \sigma^{2}\boldsymbol I)\), donde \(\boldsymbol 0\) es un vector con todas las entradas iguales a cero, y \(\boldsymbol I\) es la matriz identidad.
2.2 Estimación de los coeficientes de regresión
La estimación de \(\boldsymbol \beta\) se hace a través del método de mínimos cuadrados ordinarios. Por lo tanto, debemos encontrar el vector \(\widehat{\boldsymbol \beta}\) que minimice: \[ S(\boldsymbol \beta) = \sum_{i=1}^{n}\left(y_{i} - \boldsymbol x_{i}'\boldsymbol \beta\right)^2 = \sum_{i=1}^{n}e_{i}^2. \] En forma matricial, tenemos:
\[\begin{equation} \begin{split} S(\boldsymbol \beta) &= \boldsymbol e'\boldsymbol e= (\boldsymbol y- \boldsymbol X\boldsymbol \beta)'(\boldsymbol y- \boldsymbol X\boldsymbol \beta) \\ &= \boldsymbol y'\boldsymbol y- \boldsymbol \beta'\boldsymbol X'\boldsymbol y- \boldsymbol y'\boldsymbol X\boldsymbol \beta+ \boldsymbol \beta'\boldsymbol X'\boldsymbol X\boldsymbol \beta\\ &= \boldsymbol y'\boldsymbol y- 2\boldsymbol \beta'\boldsymbol X'\boldsymbol y+ \boldsymbol \beta'\boldsymbol X'\boldsymbol X\boldsymbol \beta. \end{split} \nonumber \end{equation}\] Por lo tanto, \(\widehat{\boldsymbol \beta}\) debe satisfacer: \[ \left. \frac{\partial S}{\partial \boldsymbol \beta} \right|_{\widehat{\boldsymbol \beta}} = - 2\boldsymbol X'\boldsymbol y+ 2\boldsymbol X'\boldsymbol X\widehat{\boldsymbol \beta}= \boldsymbol 0. \] A partir de aquí obtenemos las ecuaciones normales: \[ \boldsymbol X'\boldsymbol X\widehat{\boldsymbol \beta}= \boldsymbol X'\boldsymbol y. \] En más detalle:
\[\begin{gather} \begin{pmatrix} n & \sum x_{i1} & \sum_{i=1}^{n}x_{i2} & \ldots & \sum_{i=1}^{n}x_{i,p-1} \\ \sum x_{i1} & \sum x_{i1}^2 & \sum x_{i1}x_{i2} & \ldots & \sum x_{i1}x_{i,p-1} \\ \sum x_{i2} & \sum x_{i1}x_{i2} & \sum x_{i2}^2 & \ldots & \sum x_{i2}x_{i,p-1} \\ \vdots & \vdots & \vdots & \ddots & \vdots \\ \sum x_{i,p-1} & \sum x_{i1}x_{i,p-1} & \sum x_{i2}x_{i,p-1} & \ldots & \sum x_{i,p-1}^{2} \\ \end{pmatrix} \begin{pmatrix} \widehat{\beta}_{0} \\ \widehat{\beta}_{1} \\ \vdots \\ \widehat{\beta}_{p-1} \end{pmatrix} = \begin{pmatrix} \sum y_{i} \\ \sum x_{i1}y_{i} \\ \vdots \\ \sum x_{i,p-1}y_{i} \end{pmatrix} \nonumber \end{gather}\]
Por lo cual, el estimador por mínimos cuadrados es: \[ \widehat{\boldsymbol \beta}= (\boldsymbol X'\boldsymbol X)^{-1}\boldsymbol X'\boldsymbol y. \] Note que es necesario que \(\boldsymbol X\) sea de rango completo, \(\mbox{rango}(\boldsymbol X) = p \leq n\). Esta restricción es necesaria para asegurar que \(\boldsymbol X'\boldsymbol X\) sea no singular. Si \(\boldsymbol X'\boldsymbol X\) es singular, implica que existe una combinación lineal entre las columnas de \(\boldsymbol X\), o que \(\mbox{rango}(\boldsymbol X) < p\).
El valor ajustado de \(y\) para el vector de covariables \(\boldsymbol x_{i}\) es \(\widehat{y}_{i}= \boldsymbol x_{i}'\widehat{\boldsymbol \beta}\). Definiendo \(\widehat{\boldsymbol y}= (\widehat{y}_{1},\widehat{y}_{2},\ldots,\widehat{y}_{n})\), tenemos que: \[ \widehat{\boldsymbol y}= \boldsymbol X\widehat{\boldsymbol \beta}= \boldsymbol X(\boldsymbol X'\boldsymbol X)^{-1}\boldsymbol X'\boldsymbol y= \boldsymbol H\boldsymbol y. \] ] La matriz \(\boldsymbol H= \boldsymbol X(\boldsymbol X'\boldsymbol X)^{-1}\boldsymbol X'\) es llamada matriz hat (sombrero) y desempeña un papel importante en el análisis de regresión.
Los residuos del modelo \((e_{i}=y_{i}-\widehat{y}_{i})\) también se pueden expresar en forma matricial como: \[ \boldsymbol e= \boldsymbol y- \boldsymbol X'\widehat{\boldsymbol \beta}= \boldsymbol y- \boldsymbol X(\boldsymbol X'\boldsymbol X)^{-1}\boldsymbol X'\boldsymbol y= \boldsymbol y- \boldsymbol H\boldsymbol y= (\boldsymbol I_{n} - \boldsymbol H)\boldsymbol y. \]
2.2.1 Estimación de \(\sigma^{2}\)
Al igual que en la regresión simple, el estimador de \(\sigma^{2}\) es el cuadrado medio del error, definido como: \[ MS_{res} = \frac{SS_{res}}{n-p}, \] donde: \[\begin{equation} \begin{split} SS_{res} &= \sum_{i=1}^{n}e^{2}_{i} = \boldsymbol e'\boldsymbol e= (\boldsymbol y- \boldsymbol X\widehat{\boldsymbol \beta})'(\boldsymbol y- \boldsymbol X\widehat{\boldsymbol \beta}) \\ &= (\boldsymbol y- \boldsymbol H\boldsymbol y)'(\boldsymbol y- \boldsymbol H\boldsymbol y) = \boldsymbol y'(\boldsymbol I_{n}-\boldsymbol H)'(\boldsymbol I_{n}-\boldsymbol H)\boldsymbol y= \boldsymbol y'(\boldsymbol I_{n} - \boldsymbol H)\boldsymbol y. \end{split} \nonumber \end{equation}\] Se puede demostrar que \(MS_{res}\) es un estimador insesgado de \(\sigma^{2}\), es decir \(E(MS_{res})=\sigma^{2}\). Para esto debemos calcular el valor esperado de \(SS_{res}\).
Sabemos que \(E(\boldsymbol y) = \boldsymbol X\boldsymbol \beta\) y \(V(\boldsymbol y) = \sigma^{2}\boldsymbol I_{n}\), entonces: \[ E(SS_{res}) = E\left[\boldsymbol y'(\boldsymbol I_{n} - \boldsymbol H)\boldsymbol y\right] = \sigma^{2}\mbox{tr}\left( \boldsymbol I_{n} - \boldsymbol H\right) + \boldsymbol \beta'\boldsymbol X'(\boldsymbol I_{n} - \boldsymbol H)\boldsymbol X\boldsymbol \beta= (n-p)\sigma^{2}. \] Por lo tanto, \(E(MS_{res}) = E(SS_{res})/(n-p) = \sigma^{2}\).
2.2.2 Grasa corporal - estimación de parámetros
Para ajustar el modelo:
\[
\mbox{brozek}_{i} = \beta_{0} + \beta_{1}\mbox{abdom}_{i} + \beta_{2}\mbox{adipos}_{i}+ \beta_{3}\mbox{age}_{i} + \varepsilon_{i},
\]
donde \(\varepsilon_{i}\sim N(0,\sigma^{2})\), y \(cov(\varepsilon_{j},\varepsilon_{k})=0\) para todo \(j\neq k\), usamos la función lm de R:
##
## Call:
## lm(formula = brozek ~ abdom + adipos + age, data = fat)
##
## Coefficients:
## (Intercept) abdom adipos age
## -37.13370 0.63498 -0.21192 0.05996De aquí temenos que: \[ E(\mbox{brozek} | \mbox{abdom}, \mbox{weight},\mbox{age})= -37.134+0.635\mbox{abdom}-0.212\mbox{adipos} + 0.060\mbox{age}. \] Aquí vemos que la circunferencia del abdomen y la edad tienen un efecto positivo sobre el % de grasa corporal. Sin embargo, el efecto del IMC es negativo. Específicamente, la diferencia media en el porcentaje de grasa corporal entre hombres que presentan una variación de un centímetro en la circunferencia del abdomen, manteniendo constantes el resto de las covariables (misma edad e IMC), es de 0.63%. De igual forma, la diferencia media en el porcentaje de grasa corporal entre hombres que difieren en un año de edad, con las demás covariables constantes, es de 0.060%. Por último, la diferencia para hombres que presentan una variación de una unidad en el IMC, manteniendo constantes las demás covariables, es de -0.212%.
Además, la estimación de \(\sigma^{2}\) es:
## [1] 11.220062.2.3 Propiedades de los estimadores por MCO
Si \(E(\boldsymbol \varepsilon)= \boldsymbol 0\), el valor esperado de \(\widehat{\boldsymbol \beta}\) es: \[\begin{equation} \begin{split} E(\widehat{\boldsymbol \beta}) =& E\left[ (\boldsymbol X'\boldsymbol X)^{-1}\boldsymbol X'\boldsymbol y\right] = E\left[ \boldsymbol X'\boldsymbol X)^{-1}\boldsymbol X'(\boldsymbol X\boldsymbol \beta+ \boldsymbol \varepsilon) \right] \\ =& E\left[ (\boldsymbol X'\boldsymbol X)^{-1}\boldsymbol X'\boldsymbol X\boldsymbol \beta+ (\boldsymbol X'\boldsymbol X)^{-1}\boldsymbol X'\boldsymbol \varepsilon\right] = \boldsymbol \beta. \end{split} \nonumber \end{equation}\] Por lo tanto, \(\widehat{\boldsymbol \beta}\) es un estimador insesgado de \(\boldsymbol \beta\).
Si \(V(\boldsymbol \varepsilon)= \sigma^2\boldsymbol I\), la matriz de varianzas-covarianzas de \(\widehat{\boldsymbol \beta}\) es: \[\begin{equation} \begin{split} V(\widehat{\boldsymbol \beta}) &= V\left[ (\boldsymbol X'\boldsymbol X)^{-1}\boldsymbol X'\boldsymbol y\right] = (\boldsymbol X'\boldsymbol X)^{-1}\boldsymbol X'V(\boldsymbol y)\boldsymbol X(\boldsymbol X'\boldsymbol X)^{-1} \\ &= \sigma^{2}(\boldsymbol X'\boldsymbol X)^{-1}\boldsymbol X'\boldsymbol X(\boldsymbol X'\boldsymbol X)^{-1} = \sigma^{2}(\boldsymbol X'\boldsymbol X)^{-1}. \end{split} \nonumber \end{equation}\] Si \(\boldsymbol C=(\boldsymbol X'\boldsymbol X)^{-1}\), entonces \(V(\widehat{\beta}_{j}) = \sigma^{2}c_{jj}\) y \(Cov(\widehat{\beta}_{j},\widehat{\beta}_{k})=\sigma^{2}c_{jk}\), donde \(c_{jk}\) es la entrada \((j,k)\) de la matriz \(\boldsymbol C\).
Teorema de Gauss-Markov
Si, \(E(\boldsymbol \varepsilon) = \boldsymbol 0\) y \(V(\boldsymbol \varepsilon) = \sigma^{2}\boldsymbol I_{n}\), el estimador por MCO, \(\widehat{\boldsymbol \beta}= (\boldsymbol X'\boldsymbol X)\boldsymbol X'\boldsymbol y\), es el mejor estimador lineal insesgado de \(\boldsymbol \beta\). Esto quiere decir que es el estimador con menor varianza entre la clase de estimador insesgados que son combinaciones lineales de \(y\). Para la demostración, ver Sección C4 de Montgomery, Peck, and Vining (2012).
Además, si \(\boldsymbol \varepsilon\sim N(\boldsymbol 0, \sigma^{2}\boldsymbol I_{n})\), el estimador por MCO coincide con el estimador por máxima verosimilitud.
2.3 Pruebas de hipótesis
Después de estimar el modelo podemos preguntarnos: - ¿el modelo hace un buen ajuste de los datos? - ¿cuales regresores específicos parecen importantes?
Para resolver estas preguntas podemos realizar pruebas de hipótesis. Generalmente, estos test requieren que \(\boldsymbol \varepsilon\sim N(\boldsymbol 0,\sigma^{2}\boldsymbol I_{n})\).
2.3.1 Análisis de varianza
Para probar la significancia del modelo (determinar si que la relación entre \(y\) y algunas de las covariables es lineal) se plantean las siguientes hipótesis: \[\begin{equation} \begin{split} H_{0}:& \beta_{1}=\beta_{2}=\ldots=\beta_{p-1} = 0 \\ H_{1}:& \beta_{j}\neq 0 \mbox{ para al menos un }j. \end{split} \tag{2.3} \end{equation}\] El rechazo de esta hipótesis nula implica que al menos uno de los regresores \(x_1, x_2,\ldots , x_{p-1}\) contribuye significativamente al modelo.
Igual que en la regresión simple, el estadístico de prueba se encuentra a partir de la partición de la suma de cuadrados totales: \[ SS_{T} = SS_{R} + SS_{res}, \] donde: - \(SS_{R} = \sum_{i=1}^{n}(\widehat{y}_{i}-\bar{y})^{2} = (\boldsymbol H\boldsymbol y- \frac{1}{n}\boldsymbol 1\boldsymbol 1'\boldsymbol y)'(\boldsymbol H\boldsymbol y- \frac{1}{n}\boldsymbol 1\boldsymbol 1'\boldsymbol y)=\boldsymbol y'(\boldsymbol H- \frac{1}{n}\boldsymbol 1\boldsymbol 1')\boldsymbol y\), - \(SS_{res} = \sum_{i=1}^{n}(y_{i}-\widehat{y}_{i})^{2} = (\boldsymbol y- \boldsymbol H\boldsymbol y)'(\boldsymbol y- \boldsymbol H\boldsymbol y)=\boldsymbol y'(\boldsymbol I_n-\boldsymbol H)\boldsymbol y\),
y \(\boldsymbol 1\) es un vector cuyas entradas son iguales a \(1\).
Se tiene que, \(\frac{SS_{R}}{\sigma^2} \sim \chi^2_{r,\lambda}\), con: \[\begin{equation} \lambda = \frac{\boldsymbol \beta^{*'}\boldsymbol X_{c}'\boldsymbol X_{c}\boldsymbol \beta^{*}}{\sigma^{2}}, \tag{2.4} \end{equation}\] donde donde \(\boldsymbol \beta^{*} = (\beta_{1},\beta_{2},\ldots,\beta_{p-1})'\) y \[ \boldsymbol X_{c} = \begin{pmatrix} x_{11} - \bar{x}_{1} & x_{12} - \bar{x}_{2} & \ldots & x_{1,p-1} - \bar{x}_{p-1} \\ x_{21} - \bar{x}_{1} & x_{22} - \bar{x}_{2} & \ldots & x_{2,p-1} - \bar{x}_{p-1} \\ \vdots & \vdots & \ddots & \vdots \\ x_{i1} - \bar{x}_{1} & x_{i2} - \bar{x}_{2} & \ldots & x_{i,p-1} - \bar{x}_{p-1} \\ \vdots & \vdots & \ddots & \vdots \\ x_{n1} - \bar{x}_{1} & x_{n2} - \bar{x}_{2} & \ldots & x_{n,p-1} - \bar{x}_{p-1} \\ \end{pmatrix}. \] De igual forma, \(\frac{SS_{res}}{\sigma^2} \sim \chi^2_{n-p}\). Finalmente, se puede probar que \(SS_{R}\) y \(SS_{res}\) son independientes.
Si \(H_{0}\) es cierta, tenemos que: \[ \frac{SS_{res}}{\sigma^{2}}\sim\chi^{2}_{n-p} \mbox{ y } \frac{SS_{R}}{\sigma^{2}} \sim \chi^{2}_{p-1}. \] Por lo tanto, \[ F_{0} = \frac{SS_{R}/(p-1)}{SS_{res}/(n-p)} = \frac{MS_{R}}{MS_{res}} \sim F_{p-1,n-p}. \]
Si \(H_{0}\) no es cierta, tenemos que \(F_{0}\) sigue una distribución \(F\) no-central con \(p-1\) y \(n-p\) grados de libertad y parámetro de no centralidad (2.4).
Estos resultados nos indican que si el valor \(F_{0}\) es grande, entonces al menos un \(\beta_{j}\) es diferente de cero.
Por lo tanto, para probar las hipótesis (2.3) calculamos el estadístico de prueba \(F_{0} = \frac{MS_{R}}{MS_{res}}\), y rechazamos \(H_{0}\) si \(F_{0} > F_{1-\alpha,p-1,n-p}\).
A partir de las sumas de cuadrados podemos calcular el coeficiente de determinación: \[ R^{2} = 1-\frac{SS_{res}}{SS_{T}}. \] A medida que agregamos mas covariables al modelo el \(R^{2}\) aumenta (o permanece igual), sin importar si la covariable agregada tiene una contribución importante en el ajuste. Esto hace que sea difícil determinar si el incremento en el \(R^{2}\) al agregar una covariable sea relevante. Por esta razón, también podemos usar el coeficiente de determinación ajustado: \[ R^{2}_{adj} = 1-\frac{SS_{res}/(n-p)}{SS_{T}/(n-1)} = 1 - \frac{MS_{res}}{SS_T/(n-1)}. \] Aunque no tiene interpretación, el \(R^{2}_{adj}\) puede usarse para comparar modelos al agregar nuevas covariables. Dado que \(SS_{T}/(n-1)\) es constante, el \(R^{2}_{adj}\) solo aumentará al agregar una covariable nueva al modelo si la adición de la covariable reduce el \(MS_{res}\).
2.3.2 Pruebas individuales sobre los coeficientes
Al rechazar \(H_{0}\) de la prueba de hipótesis (2.3) concluimos que al menos un coeficiente es diferente de cero. Por lo tanto, una o más covariables tienen un aporte significativo en el modelo. El paso que sigue es identificar estas covariables relevantes.
Para esto podemos plantear las siguientes hipótesis individuales sobre los coeficientes del modelo: \[ H_{0}: \beta_{j} = 0 \qquad H_{1}: \beta_{j} \neq 0. \] El estadística de prueba es: \[ t_{0} = \frac{\widehat{\beta}_{j}}{se(\widehat{\beta}_{j})} = \frac{\widehat{\beta}_{j}}{\sqrt{MS_{res}c_{jj}}}, \] donde \(c_{jj}\) es la entrada \((j,j)\) de la matriz \(\boldsymbol C= (\boldsymbol X'\boldsymbol X)^{-1}\). Rechazamos \(H_{0}\) si \(|t_{0}| > t_{1-\alpha/2,n-p}\).
Este es una prueba parcial, puesto que estamos evaluando la significancia de \(x_{j}\) cuando las demás covariables \(x_{k}\), para \(k\neq j\), ya están incluidas en el modelo. Por lo tanto, si no rechazamos \(H_{0}\), podemos concluir que, cuando los demás regresores están en el modelo, la covariable \(x_{j}\) no tiene un aporte significativo.
2.3.3 Pruebas sobre subconjuntos de coeficientes
Para probar la significancia de un subconjunto de coeficientes del modelo hacemos uso de la suma de cuadrados extra. Primero, consideremos el siguiente modelo de regresión: \[ \boldsymbol y= \boldsymbol X\boldsymbol \beta+ \boldsymbol \varepsilon, \] donde \(\boldsymbol X\) es una matrix \(n \times p\) y \(\boldsymbol \beta\) es el vector de coeficientes de longitud \(p\). Queremos probar si un subconjunto \(r < p\) de covariables tienen un aporte significativo en el modelo. Para esto hacemos la siguiente partición del vector \(\boldsymbol \beta\): \[ \boldsymbol \beta= \begin{pmatrix} \beta_{0} \\ \beta_{1} \\ \vdots \\ \beta_{p-r-1} \\ \hline \beta_{p-r} \\ \beta_{p-r+1} \\ \vdots \\ \beta_{p} \end{pmatrix} = \begin{pmatrix} \boldsymbol \beta_{1} \\ \hline \boldsymbol \beta_{2}\end{pmatrix}, \] donde \(\boldsymbol \beta_{1}\) y \(\boldsymbol \beta_{2}\) son vector de dimensión \((p-r)\) y \((r)\), respectivamente. Por lo tanto, queremos realizar la siguiente prueba de hipótesis: \[\begin{equation} H_{0}: \boldsymbol \beta_{2} = \boldsymbol 0\qquad H_{1}: \boldsymbol \beta_{2} \neq \boldsymbol 0. \tag{2.5} \end{equation}\] El modelo anterior se puede re-escribir de la siguiente forma: \[ \boldsymbol y= \boldsymbol X\boldsymbol \beta+ \boldsymbol \varepsilon= \boldsymbol X_{1}\boldsymbol \beta_{1}+ \boldsymbol X_{2}\boldsymbol \beta_{2} + \boldsymbol \varepsilon, \] donde \(\boldsymbol X_{1}\) es la matriz \(n\times (p-r)\) que contiene las columnas de \(\boldsymbol X\) asociadas con \(\boldsymbol \beta_{1}\), y \(\boldsymbol X_{2}\) es la matriz \(n\times r\) que contiene las columnas de \(\boldsymbol X\) asociadas con \(\boldsymbol \beta_{2}\). Este es llamado el modelo completo.
Para el modelo completo tenemos:
- Estimador de \(\boldsymbol \beta\): \[ \widehat{\boldsymbol \beta}= (\boldsymbol X'\boldsymbol X)^{-1}\boldsymbol X'\boldsymbol y. \]
- Suma de cuadrados del modelo: \[ SS_{R}(\boldsymbol \beta) = \boldsymbol y'(\boldsymbol H- \frac{1}{n}\boldsymbol 1\boldsymbol 1')\boldsymbol y. \]
- Cuadrado medio del error: \[ MS_{res} = \frac{\boldsymbol y'(\boldsymbol I- \boldsymbol H)\boldsymbol y}{n-p}. \]
Para evaluar la contribución de los regresores asociados a \(\boldsymbol \beta_{2}\), ajustamos el modelo asumiendo que \(H_{0}\) es cierta. De esta forma tenemos el modelo reducido: \[ \boldsymbol y= \boldsymbol X_{1}\boldsymbol \beta_{1} + \boldsymbol \varepsilon. \] Para el modelo reducido tenemos:
- Estimador de \(\boldsymbol \beta_{1}\): \[ \widehat{\boldsymbol \beta}_{1} = (\boldsymbol X_{1}'\boldsymbol X_{1})^{-1}\boldsymbol X_{1}'\boldsymbol y. \]
- Suma de cuadrados del modelo: \[ SS_{R}(\boldsymbol \beta_{1}) = \boldsymbol y'(\boldsymbol H_1 - \frac{1}{n}\boldsymbol 1\boldsymbol 1')\boldsymbol y, \] con \(\boldsymbol H_1 = \boldsymbol X_1(\boldsymbol X_1'\boldsymbol X_1)^{-1}\boldsymbol X_1'\).
Entonces, la suma de cuadrados debido a \(\boldsymbol \beta_{2}\) dado que \(\boldsymbol \beta_{1}\) ya está en el modelo es: \[ SS_{R}(\boldsymbol \beta_{2}| \boldsymbol \beta_{1}) = SS_{R}(\boldsymbol \beta) - SS_{R}(\boldsymbol \beta_{1}), \] Esta suma de cuadrados es llamada la suma de cuadrados extra debido a \(\boldsymbol \beta_2\) puesto que mide el incremento en la suma de cuadrados de la regresión como resultado de adicionar los regresores \(\boldsymbol X_{2}\) en el modelo que ya contiene \(\boldsymbol X_{1}\).
Se puede probar que \(SS_{R}(\boldsymbol \beta_{2}| \boldsymbol \beta_{1})\) y \(MS_{res}\) son independientes. Por lo tanto, podemos utilizar el siguiente estadístico de prueba: \[ F_{0} = \frac{SS_{R}(\boldsymbol \beta_{2}|\boldsymbol \beta_{1})/r}{MS_{res}}. \] Si \(H_{0}\) es cierta entonces \(F_{0} \sim F_{r,n-p}\). Si \(H_{0}\) no es cierta, entonces \(F_{0}\) sigue una distribución \(F\) no-central con parámetro de no centralidad igual a: \[ \lambda = \frac{1}{\sigma^{2}}\boldsymbol \beta_{2}'\boldsymbol X_{2}'\left[ \boldsymbol I_{n} - \boldsymbol X_{1}(\boldsymbol X_{1}'\boldsymbol X_{1})^{-1}\boldsymbol X_{1}'\right]\boldsymbol X_{2}\boldsymbol \beta_{2}. \] Note que si hay una relación casi colineal entre \(\boldsymbol X_{1}\) y \(\boldsymbol X_{2}\) (multicolinealidad), \(\lambda\) es cercano a cero pesar que \(\boldsymbol \beta_{2}\) sea marcadamente distinto de cero. Es decir, que la prueba tiene poca capacidad de indicar diferencias (poco poder) en presencia de multicolinealidad. Caso contrario, el máximo poder se alcanza cuando \(\boldsymbol X_{1}\) y \(\boldsymbol X_{2}\) son ortogonales (es decir \(\boldsymbol X_{2}'\boldsymbol X_{1} = \boldsymbol 0\)).
Entonces, si \(F_{0} > F_{1-\alpha,r,n-p}\) rechazamos \(H_{0}\) y concluimos que al menos un coeficiente en \(\boldsymbol \beta_{2}\) es diferente de cero. Consecuentemente, al menos una de las covariables en \(\boldsymbol X_{2}\) tiene un aporte significativo dentro del modelo.
Ejemplo
Considere el modelo: \[ y_{i} = \beta_{0} + \beta_{1}x_{i1} + \beta_{2}x_{i2} + \beta_{3}x_{i3} + \varepsilon_{i}. \] La suma de cuadrados del modelo se puede descomponer de la siguiente forma: \[ SS_{R}=SS_{R}(\beta_{1},\beta_{2},\beta_{3}| \beta_{0}) = SS_{R}(\beta_{1}|\beta_{0}) + SS_{R}(\beta_{2}|\beta_{0},\beta_{1}) + SS_{R}(\beta_{3}|\beta_{0},\beta_{1},\beta_{2}), \] donde cada suma de cuadrados en el lado derecho tiene un grado de libertad. Además, el order de los regresores en estos componentes marginales es arbitrario. Por lo que la siguiente descomposición alternativa es también válida: \[ SS_{R}(\beta_{1},\beta_{2},\beta_{3}| \beta_{0})=SS_{R}(\beta_{2}|\beta_{0}) + SS_{R}(\beta_{3}|\beta_{0},\beta_{2}) + SS_{R}(\beta_{1}|\beta_{0},\beta_{2},\beta_{3}). \]
Sin embargo, la siguiente partición de la suma de cuadrados de la regrsión es generalmente inválida: \[ SS_{R}(\beta_{1},\beta_{2},\beta_{3}| \beta_{0})\neq SS_{R}(\beta_{1}|\beta_{0},\beta_{2},\beta_{3}) + SS_{R}(\beta_{2}|\beta_{0},\beta_{1},\beta_{3}) + SS_{R}(\beta_{3}|\beta_{0},\beta_{1},\beta_{2}). \]
2.4 Prueba de hipótesis lineal general
Suponga que estamos interesados en las siguientes hipótesis: \[\begin{equation} H_{0}: \boldsymbol T\boldsymbol \beta=\boldsymbol 0\qquad H_{1}: \boldsymbol T\boldsymbol \beta\neq \boldsymbol 0, \tag{2.6} \end{equation}\] donde \(\boldsymbol T\) es una matriz \(m \times p\) de constantes, tal que \(r\) de las \(m\) ecuaciones de \(\boldsymbol T\boldsymbol \beta=\boldsymbol 0\) son independientes.
El modelo completo (FM) es: \[ \boldsymbol y=\boldsymbol X\boldsymbol \beta+\boldsymbol \varepsilon, \] El estimador de \(\boldsymbol \beta\) es \(\widehat{\boldsymbol \beta}= (\boldsymbol X'\boldsymbol X)^{-1}\boldsymbol X'\boldsymbol y\), y la suma de cuadrados de los residuos es \(SS_{res}(FM)\) (con \(n-p\) grados de libertad).
El modelo reducido (RM) se obtiene al resolver las \(r\) ecuaciones independientes de \(\boldsymbol T\boldsymbol \beta= \boldsymbol 0\) para los \(r\) coeficientes en el modelo completo en términos de los \(p-r\) coeficientes restantes. Esto lleva al siguiente RM: \[ \boldsymbol y=\boldsymbol Z\boldsymbol \gamma+\boldsymbol \varepsilon, \] donde \(\boldsymbol Z\) es una matriz \(n\times (p-r)\) y \(\boldsymbol \gamma\) es un vector de dimiensión \((p-r)\) de coeficientes de regresión. La suma de cuadrados de los residuos de este modelo es \(SS_{res}(RM)\) (con \(n-p+r\) grados de libertad).
Dado que el modelo reducido tiene menos parámetros que el modelo completo, \(SS_{res}(RM) \geq SS_{res}(FM)\). Para probar (2.6) usamos la diferencia entre las sumas de cuadrados de los residuos: \[ SS_{H} = SS_{res}(RM) - SS_{res}(FM), \] con \(r\) grados de libertad. \(SS_{H}\) es llamado la suma de cuadrados debido a \(H_{0}:\boldsymbol T\boldsymbol \beta=\boldsymbol c\). El estadístico de prueba es: \[ F_{0} = \frac{SS_{H}/r}{SS_{res}(FM)/(n-p)} = \frac{\widehat{\beta}'\boldsymbol T[\boldsymbol T(\boldsymbol X'\boldsymbol X)\boldsymbol T']^{-1}\boldsymbol T\widehat{\boldsymbol \beta}/r}{SS_{res}(FM)/(n-p)}. \] Rechazamos \(H_{0}\) si \(F_{0} > F_{1-\alpha,r,n-p}\).
La hipótesis anterior se puede generalizar de la siguiente forma: \[\begin{equation} H_{0}: \boldsymbol T\boldsymbol \beta=\boldsymbol c\qquad H_{1}: \boldsymbol T\boldsymbol \beta\neq \boldsymbol c, \tag{2.7} \end{equation}\] Para este caso, el estadístico de prueba es: \[ F_{0} = \frac{(\boldsymbol T\widehat{\boldsymbol \beta}-\boldsymbol c)'[\boldsymbol T(\boldsymbol X'\boldsymbol X)\boldsymbol T']^{-1}(\boldsymbol T\widehat{\boldsymbol \beta}-\boldsymbol c)/r}{SS_{res}(FM)/(n-p)}. \] Si \(H_{0}\) es cierta, \(F_{0}\sim F_{r,n-p}\). Por lo tanto, rechazamos \(H_{0}\) si \(F_{0} > F_{1-\alpha,r,n-p}\).
ejemplo
Considere el modelo: \[ y_{i} = \beta_{0} + \beta_{1}x_{i1}+ \beta_{2}x_{i2}+ \beta_{3}x_{i3}+ \beta_{3}x_{i3} + \varepsilon_{i}, \] y queremos probar las siguientes hipótesis: \[\begin{align*} H_{0}:& \beta_{1}=0 & H_{1}:&\beta_{1}\neq 0 \\ & 2\beta_{2}-\beta_{3}=3 & &2\beta_{2}-\beta_{3}\neq 3 \\ \end{align*}\] De aquí tenemos que: \[ \boldsymbol T= \begin{pmatrix} 1 & 0 & 0 & 0 \\ 0 & 2 & -1 & 0 \\ \end{pmatrix} \mbox{ y } \boldsymbol c= \begin{pmatrix} 0 \\ 3 \end{pmatrix}. \] Si no rechazamos \(H_{0}\), podríamos estimar \(\boldsymbol \beta\) sujo a las restricciones impuestar por la hipótesis nula (usando mínimos cuadrados restringidos).
Grasa corporal - pruebas de hipótesis
Los resultados de las pruebas de hipótesis individuales sobre los coeficientes, análisis de varianza y coeficientes de determianción se obtiene a partir del resumen del modelo:
##
## Call:
## lm(formula = brozek ~ abdom + adipos + age, data = fat)
##
## Residuals:
## Min 1Q Median 3Q Max
## -15.5026 -3.1891 0.1558 3.4636 12.3348
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -37.13370 2.54301 -14.602 <2e-16 ***
## abdom 0.63498 0.07177 8.847 <2e-16 ***
## adipos -0.21192 0.20790 -1.019 0.3090
## age 0.05996 0.02367 2.533 0.0119 *
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 4.449 on 248 degrees of freedom
## Multiple R-squared: 0.6744, Adjusted R-squared: 0.6705
## F-statistic: 171.2 on 3 and 248 DF, p-value: < 2.2e-16A partir de estos resutados tenemos que \(F_{0}=171.2252\) con un valor-\(p\) asociado de \(0.000\), es decir que al menos uno de los coeficientes de regresión es diferente de cero. Además, el \(67\)% de la variabilidad del % de grasa corporal es explicada por la circunferencia del abdomen, edad e IMC \((R^{2}=0.6744)\). Note que hubo un aumento leve en el \(R^{2}\) respecto al modelo que solo incluye la circunferencia del abdomen como covariable (\(R^{2} = 0.662\)).
A partir de las pruebas de hipótesis individuales, podemos decir que el IMC no tiene un aporte significativo cuando el modelo ya incluye las covariables circunferencia del abdomen y la edad (\(t_{0}=-1.02\) con un valor-\(p\) asociado de 0.309). Por otro lado, los otros dos efectos son significativos.
Ahora consideremos un modelo ingresando dos covariables más, circunferencia de los muslos (thigh) y las caderas (hip):
\[
\mbox{brozek}_{i} = \beta_{0} + \beta_{1}\mbox{age}_{i} + \beta_{2}\mbox{abdom}_{i} + \beta_{3}\mbox{adipos}_{i} + \beta_{4}\mbox{thigh}_{i} + \beta_{5}\mbox{hip}_{i} + \varepsilon_{i},
\]
donde \(\varepsilon_{i} \sim N(0,\sigma^{2})\) y \(cov(\varepsilon_{i},\varepsilon_{j})=0\). El resumen del modelo ajustado es:
##
## Call:
## lm(formula = brozek ~ age + abdom + adipos + thigh + hip, data = fat)
##
## Residuals:
## Min 1Q Median 3Q Max
## -11.2165 -2.9719 -0.2144 3.0470 11.9142
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -18.32479 5.63905 -3.250 0.00132 **
## age 0.02522 0.02829 0.891 0.37367
## abdom 0.78438 0.07938 9.882 < 2e-16 ***
## adipos 0.00249 0.21850 0.011 0.99092
## thigh 0.18915 0.12923 1.464 0.14455
## hip -0.47813 0.11705 -4.085 5.97e-05 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 4.316 on 246 degrees of freedom
## Multiple R-squared: 0.696, Adjusted R-squared: 0.6899
## F-statistic: 112.7 on 5 and 246 DF, p-value: < 2.2e-16Respecto al modelo anterior, hay un aumento del \(R^{2}\) y el \(R^{2}_{adj}\). Por lo cuál podemos concluir que al ingresar estas covariables el ajuste mejoró. Aunque, varios de los efectos no son significativos a partir de las pruebas individuales. Esto no necesariamente quiere decir que podemos eliminar estas covariables del modelo, recordemos que las pruebas \(t\) son condicionales (se evalúa el efecto de la covariable cuando el modelo ya incluye las restantes). Para determinar si podemos eliminar esas tres covariables del modelo, realizamos la siguiente prueba de hipótesis:
\[
H_{0}: \beta_{1} = \beta_{3} = \beta_{4} = 0 \qquad H_{0}: \beta_{j} \neq 0 \mbox{ para algún }j=1,3,4.
\]
En R podemos hacer esto a través de la función anova:
## Analysis of Variance Table
##
## Model 1: brozek ~ abdom + hip
## Model 2: brozek ~ age + abdom + adipos + thigh + hip
## Res.Df RSS Df Sum of Sq F Pr(>F)
## 1 249 4627.5
## 2 246 4583.3 3 44.209 0.7909 0.4999Aquí vemos que \(F_{0}= 0.791\) con un valor-\(p\) asociado de \(0.4999\). Por lo tanto, no tenemos evidencia suficiente para rechazar \(H_{0}\) y podemos considerar que estas tres covariables no tienen un aporte significativo sobre el % de grasa corporal cuando ya se tienen en cuenta la circunferencia del abdomen y las caderas.
2.5 Intervalos de confianza
Al igual que en caso del modelo lineal simple, también podemos hacer estimaciones por intervalos de confianza para los coeficientes del modelo, valor esperado de \(Y\) y observaciones futuras.
Para que los intervalos de confianza sean válidos se requiere que se cumplan todos los supuestos del modelo, esto es \(\boldsymbol \varepsilon\sim N(\boldsymbol 0, \sigma^{2}\boldsymbol I_{n})\).
2.5.1 Intervalos de confianza para \(\beta_{j}\)
El intervalo de confianza de \(\beta_{j}\) parte de: \[ \frac{\widehat{\beta}_{j} - \beta_{j}}{\sqrt{V(\widehat{\beta}_{j})}} = \frac{\widehat{\beta}_{j} - \beta_{j}}{\sqrt{MS_{res}c_{jj}}} \sim t_{n-p}, \] donde \(c_{jj}\) es la entrada \((j,j)\) de la matriz \(\boldsymbol C=(\boldsymbol X'\boldsymbol X)^{-1}\). Entonces, el intervalo del \((1-\alpha)100\%\) de confianza para \(\widehat{\beta}_{j}\) es: \[ \widehat{\beta}_{j} \pm t_{1-\alpha/2,n-p}\sqrt{MS_{res}c_{jj}}. \]
2.5.2 Intervalos de confianza para el valor esperado de \(Y\) y una observación futura
Ahora queremos construir un intervalo de confianza para la respuesta media de \(Y\) para un punto particular \(\boldsymbol x_{0}=(1,x_{01},x_{02},\ldots,x_{0,p-1})\). La estimación puntual en \(\boldsymbol x_{0}\) es: \[ \widehat{\mu}_{Y|\boldsymbol x_0} = \boldsymbol x_{0}'\widehat{\boldsymbol \beta}. \] Además, tenemos que \(\widehat{\mu}_{Y|\boldsymbol x_0} \sim N[\boldsymbol x_{0}'\boldsymbol \beta, V(\widehat{\mu}_{Y|\boldsymbol x_0})]\) con: \[ V(\widehat{\mu}_{Y|\boldsymbol x_0}) = \sigma^{2}\boldsymbol x_{0}'(\boldsymbol X'\boldsymbol X)^{-1}\boldsymbol x_{0}. \] Por lo tanto, el intervalo del \((1-\alpha)100\%\) de confianza para \(E(Y|\boldsymbol x_{0})\) es: \[ \widehat{\mu}_{Y|\boldsymbol x_0} \pm t_{1-\alpha/2,n-p}\sqrt{MS_{res}\boldsymbol x_{0}'(\boldsymbol X'\boldsymbol X)^{-1}\boldsymbol x_{0}}. \] De igual forma, el intervalo del \((1-\alpha)100\%\) de confianza para una observación futura en \(\boldsymbol x_{0}\) es: \[ \widehat{\mu}_{Y|\boldsymbol x_0} \pm t_{1-\alpha/2,n-p}\sqrt{MS_{res}\left[1+ \boldsymbol x_{0}'(\boldsymbol X'\boldsymbol X)^{-1}\boldsymbol x_{0}\right]}. \]
Grasa corporal - intervalos de confianza
Siguiendo con el modelo inicial, \(\mbox{brozek}_{i}=\beta_{0}+\beta_{1}\mbox{abdom}_{i}+\beta_{2}\mbox{age}_{i}+\beta_{3}\mbox{adipos}_{i}+\varepsilon_{i}\), los intervalos del 95% de confianza para los coeficientes son:
## 2.5 % 97.5 %
## (Intercept) -42.14234572 -32.1250456
## abdom 0.49362813 0.7763410
## adipos -0.62140288 0.1975599
## age 0.01334105 0.1065751La diferencia en la media del % de grasa corporal de hombres que tienen una diferencia de un centímetro en la circunferencia del abdomen, con igual valor de las demás covariables, está entre \(0.494\)% y \(0.776\)% con un nivel de confianza del 95%. Note que el intervalo de confianza para el coeficiente asociado al IMC contiene el valor \(0\) (recordemos que no rechazamos la hipótesis nula de la prueba \(t\) sobre \(\beta_{2}\)).
Queremos determinar el % de grasa corporal medio de hombres de 42 años, una circunferencia del abdomen de 89cm y un IMC de 24. Para esto podemos contruir un intervalo del 95% de confianza:
## fit lwr upr
## 1 16.81205 16.20386 17.42025Por lo tanto, la media del % de grasa corporal de hombres con estas características está entre \(16.2\)% y \(17.42\)% con un nivel de confianza del 95%.
Ahora, estamos interesados en predecir el % de grasa corporal de un hombre de \(38\) años, circunferencia del abdomen de 95cm y un IMC de 27. Entonces, debemos construimos un intervalo del 95% de predicción:
## fit lwr upr
## 1 19.74636 10.95485 28.537882.6 Extrapolación oculta en regresión múltiple
Al igual que en regresión simple, al estimar la media una nueva respuesta en un punto dado \(\boldsymbol x_{0}\) se debe tener cuidado de no extrapolar fuera de la región de los valores de las covariables observadas. En regresión múltiple es fácil extrapolar inadvertidamente, puesto que la región que contiene los datos está definida de forma conjunta por los valores que toman las covariables y no por el rango individual de cada covariable.
La Figura 2.2 muestra un ejemplo de extrapolación en el caso de un modelo de regresión con dos covariables. Se quiere hacer una predicción en el punto \((x_{01},x_{02})\) que está dentro del rango de ambos regresores, pero que fuera de la región conjunta de los datos (región roja en la figura). Por lo tanto, al realizar la predicción en este punto estaríamos extrapolando.
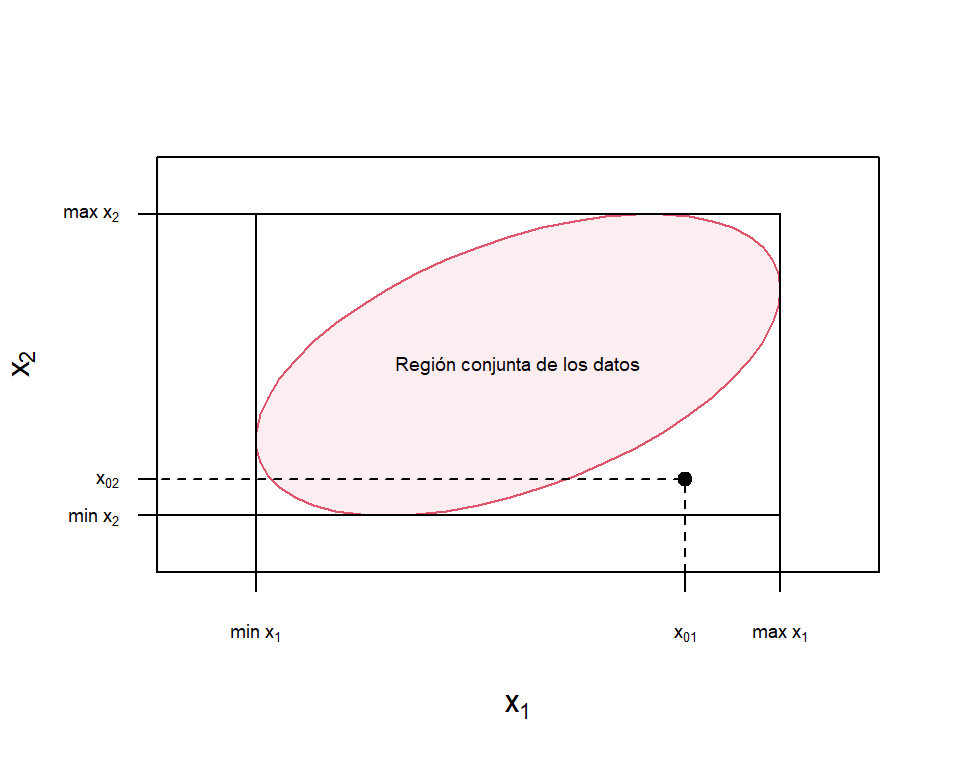
Figure 2.2: Ejemplo de extrapolación en regresión múltiple
Determinar la región conjunta de los datos en regresión múltiple no es fácil, lo que dificulta saber si se está extrapolando a la hora de hacer de una estimación. Por lo tanto, se ha propuesto determinar la región conjunta de los datos a partir del conjunto convexo mínimo que contiene todos los \(n\) datos originales, \((x_{i1}; x_{i2},\ldots,x_{i,p-1})\), para \(i = 1,2,\ldots,n\), como la envolvente de las covariables (RVH). Entonces, si un punto \((x_{01},x_{02}, \ldots,\ldots, x_{0,p-1})\) está dentro o en la frontera de la RVH, una prediccón o una estimación implica interpolación, mientras que si está fuera de la RVH, se está extrapolando.
Una aproximación de la RVH es a través de la matriz \(\boldsymbol H\). El conjunto de puntos \(\boldsymbol x\) que satisfacen, \(\boldsymbol x'(\boldsymbol X'\boldsymbol X)^{-1}\boldsymbol x\leq \max(h_{ii})\), producen un elipsoide que encierra todos los puntos dentro de la \(RVH\). Entonces, un punto \(\boldsymbol x_{0}\) está fuera de la RVH si \(h_{00} > \max{h_{ii}}\), donde: \[ h_{00} = \boldsymbol x'_{0}(\boldsymbol X'\boldsymbol X)^{-1}\boldsymbol x_{0}. \]
Grasa corporal - interpolación
Como ejemplo, consideremos el modelo con solo dos covariables: \[ \mbox{brozek}_{i} = \beta_{0} + \beta_{1}\mbox{abdom}_{i} + \beta_{5}\mbox{hip}_{i} + \varepsilon_{i}, \] donde \(\varepsilon_{i} \sim N(0,\sigma^{2})\) y \(cov(\varepsilon_{i},\varepsilon_{j})=0\). Suponga que se quiere hacer una estimación para hombres con las características que muestra la Tabla 2.1.
| 1 | 2 | 3 | |
|---|---|---|---|
| circunferencia del abdomen (cm) | 93 | 80 | 110 |
| circunferencia de la cadera (cm) | 100 | 120 | 115 |
La Figura 2.3 muestra el gráfico de dispersión de las covariables, donde los puntos rojos indican los valores donde se quieren hacer las estimaciones Aquí vemos que en los puntos \(\boldsymbol x_{01}\) y \(\boldsymbol x_{03}\) no estaríamos extrapolando. Sin embargo, el punto \(\boldsymbol x_{02}\) está alejado de la nube de puntos (aunque el punto está dentro del rango de cada covariable). No se obtuvieron observaciones de hombres con características como está. Ahora, vamos a calcular las aproximaciones de la RVH y verificar si en estos puntos estaríamos extrapolando.
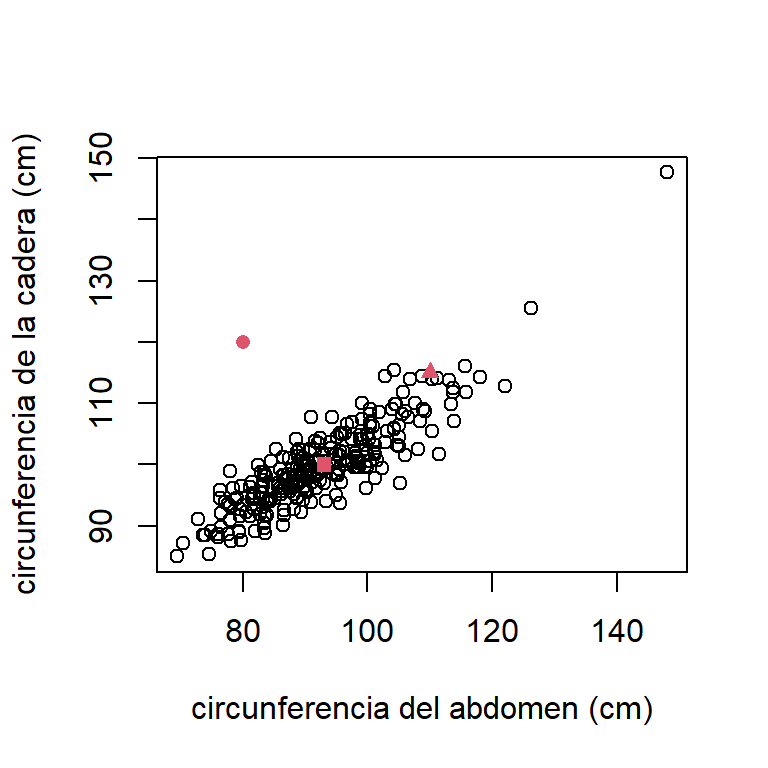
Figure 2.3: Grasa corporal. Gráfico de dispersion de la circunferencia del abdomen y la cadera. Los puntos donde se quiere hacer las estimaciones están en rojo.
modelo = lm(brozek~abdom+hip,data=fat)
newPoints = cbind(x0=1,x1=c(93,80,110),x2=c(100,120,115))
X = model.matrix(modelo)
XtX.inv = solve(t(X)%*%X)
h.values = hatvalues(modelo)
hmax = max(h.values)
h0 = apply(newPoints,1,function(x){t(x)%*%XtX.inv%*%x})
h0 >hmax## [1] FALSE TRUE FALSE2.6.1 Coeficientes normalizados de regresión
Los coeficientes de regresión están influenciados por las unidades de medida de las covariables. Exactamente las unidades de medida de $\beta_{j}$ es:\[ \frac{\mbox{la unidad de medida de }y }{\mbox{la unidad de medida de }x_{j}}. \] Dado que, por lo general, las covariables están medidas en unidades diferentes, la comparación de los coeficientes es complicada. Por esta razón, en algunas ocasiones es útil escalar los valores de las covariables y la respuesta para calcular los coeficientes de regresión adimensionales. Hay varias formas de hacer este escalamiento, aquí nos centraremos en el escalamiento de longitud unitaria.
2.6.1.1 Escalamiento de longitud unitaria
Una opción es hacer un escalamiento de longitud unitaria a las covariables: \[ z_{ij} = \frac{x_{ij}-\bar{x}_j}{\sqrt{S_{jj}}}, i=1,2,\ldots,n \quad j=1,2,\ldots,p-1, \] y la variable respuesta: \[ y_{i}^{*} = \frac{y_{i}-\bar{y}}{\sqrt{SS_{T}}}, \] donde: \[ S_{jj} = \sum_{i=1}^{n}(x_{ij} - \bar{x}_{j})^{2}. \] Con estas variables transformadas, se puede ajustar el modelo: \[ y_{i}^{*} = b_{1}z_{i1} + b_{2}z_{i2} + \ldots + b_{p-1}z_{i,p-1} = \boldsymbol z_{i}'\boldsymbol b+ \boldsymbol \varepsilon. \] El estimador por MCO es: \[ \widehat{\boldsymbol b}= (\boldsymbol Z'\boldsymbol Z)^{-1}\boldsymbol Z'\boldsymbol y^{*}. \] Note que con este escalamiento, la matriz \((\boldsymbol Z'\boldsymbol Z)\) es igual a la matriz de correlación de las covariables. Esto es: \[ (\boldsymbol Z'\boldsymbol Z) = \boldsymbol R= \begin{pmatrix} 1 & r_{12} & r_{13} & \ldots & r_{1,p-1} \\ r_{12} & 1 & r_{23} & \ldots & r_{2,p-1} \\ \vdots & \vdots & \vdots & \ddots & \vdots \\ r_{1,p-1} & 1 & r_{2,p-1} & \ldots & 1 \\ \end{pmatrix}, \] donde \(r_{jk}\) es la correlación entre las covariables \(x_{j}\) y \(x_{k}\). Además, la matriz \(\boldsymbol Z'\boldsymbol y^{*}\) es el vector de correlación entre la variable respuesta y cada covariable. Esto es: \[ \boldsymbol Z'\boldsymbol y^{*} = (r_{1y},r_{2y},r_{3y},\ldots,r_{p-1,y})', \] donde \(r_{jy}\) es la correlación entre la variable respuesta y la covariable \(x_j\).Grasa corporal - coeficientes de regresión con variables escaladas
Volvamos a considera el modelo con las covariables edad, circunferencia del abdomen e IMC. Aquí las tres tienen unidades de medida, por lo cual no es trivial determinar cual tiene mayor importancia.
Para estimar los coeficientes de regresión escalados, primero debemos escalar las variables:X = model.matrix(mod)
y = fat$brozek
Z = apply(X[,-1],2,function(x){(x-mean(x))/sqrt(sum((x-mean(x))^2))})
ys = (y-mean(y))/sqrt(sum((y-mean(y))^2))Ahora procedemos a estimar el modelo con las variables escaladas:
##
## Call:
## lm(formula = ys ~ Z - 1)
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.126246 -0.025971 0.001268 0.028206 0.100449
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## Zabdom 0.88340 0.09965 8.865 <2e-16 ***
## Zadipos -0.09975 0.09766 -1.021 0.3081
## Zage 0.09749 0.03841 2.538 0.0117 *
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.03616 on 249 degrees of freedom
## Multiple R-squared: 0.6744, Adjusted R-squared: 0.6705
## F-statistic: 171.9 on 3 and 249 DF, p-value: < 2.2e-162.7 Multicolinealidad
Un problema que puede afectar enormente el ajuste de un modelo de regresión es la multicolinealidad. Este se presenta cuando hay una dependencia casi lineal entre las covariables.
Recordemos que el estimador por MCO es \(\widehat{\boldsymbol \beta}=(\boldsymbol X'\boldsymbol X)^{-1}\boldsymbol X'\boldsymbol y\). Por lo tanto es necesario que la matriz \(\boldsymbol X'\boldsymbol X\) sea no singular. En caso contrario, no es posible encontrar la inversa y las ecuaciones normales no tendrán una única solución. Cuando sucede esto se debe a que hay al menos una columna de \(\boldsymbol X\) linealmente dependiente.
En regresión se utiliza las palabras multicolinealidad cuando hay una dependencia aproximada en las columnas de \(\boldsymbol X\). Es decir que al menos una covariable puede representarse, de forma aproximada, como una relación lineal de las otras: \[ x_{ij} \approx c_{0} + c_{1}x_{i1} + \ldots + c_{j-1}x_{i,j-1} + c_{j+1}x_{i,j+1} + \ldots + + c_{p-1}x_{i,p-1}, \] para \(i=1,\ldots,n\).
Hay que aclarar que la falta de ortogonalidad no es necesariamente un inconveniente, el problema es cuando la relación lineal entre los regresores es casi perfecta, lo que provoca problemas en las inferencias que se hagan. Uno de estos problemas se ilustra a continuación con un ejemplo.
ejemplo
Considere el siguiente modelo de regresión: \[ y_{i} = \beta_{0} + \beta_{1}x_{i1} + \beta_{2}x_{i2} + \varepsilon_{i}, \mbox{ con }\varepsilon_{i}\sim N(0,\sigma^{2}), \] y se plantean dos posibles matrices de diseño: \[ \boldsymbol X_{1} = \begin{pmatrix} 1& 1 \\ 1 & 5 \\ 2 & 1 \\ 2 & 5 \\ \end{pmatrix} \mbox{ y } \boldsymbol X_{2} = \begin{pmatrix} 1& 1 \\ 1 & 2 \\ 2 & 4 \\ 2 & 5 \\ \end{pmatrix}. \] Haciendo el escalamiento de longitud unitaria a las covariables tenemos que: \[ \boldsymbol Z_{1}'\boldsymbol Z_{1} = \begin{pmatrix} 1 & 0 \\ 0 & 1 \end{pmatrix} \mbox{ y } \boldsymbol Z_{1}'\boldsymbol Z_{1} = \begin{pmatrix} 1 & 0.95 \\ 0.95 & 1 \end{pmatrix}. \] Por lo tanto, la varianza de \(\widehat{\boldsymbol b}\) para ambos casos es: \[ V(\widehat{\boldsymbol b}_{1}) = \sigma^{2}(\boldsymbol Z_{1}'\boldsymbol Z_{1})^{-1} = \sigma^{2} \begin{pmatrix} 1 & 0 \\ 0 & 1 \end{pmatrix}^{-1} = \sigma^{2} \begin{pmatrix} 1 & 0 \\ 0 & 1 \end{pmatrix}. \] y \[ V(\widehat{\boldsymbol b}_{2}) = \sigma^{2}_{0}(\boldsymbol Z_{2}'\boldsymbol Z_{2})^{-1} = \sigma^{2} \begin{pmatrix} 1 & 0.95 \\ 0.95 & 1 \end{pmatrix}^{-1} = \sigma^{2} \begin{pmatrix} 10 & 9.49 \\ 9.49 & 10 \end{pmatrix}. \] Aquí podemos ver que la varianza de \(\widehat{\boldsymbol b}_{2}\) está inflada debido a la alta correlación entre las columnas de \(\boldsymbol X_{2}\). Es 10 veces mayor que la varianza de \(\widehat{\boldsymbol b}_{1}\) (las columnas de \(\boldsymbol X_{1}\) son independientes).
En el ejemplo anterior vemos que los valores de la diagonal de la matriz \((\boldsymbol Z'\boldsymbol Z)^{-1}\) nos indican en cuanto aumenta la varianza de las estimaciones de los coeficientes debido a la multicolinealidad. Por esta razón, estos valores toman el nombre de factores de inflación de varianza (VIFs) y son uno de los indicadores para el diagnostico de este problema.
Se puede demostrar que el VIF de \(\beta_{j}\) se puede calcular como: \[ \mbox{VIF}_{j} = \frac{1}{1-R^{2}_{j}}, \] donde \(R^{2}_{j}\) es el coeficiente de determinación obtenido ajustado una regresión de \(x_{j}\) sobre las demás covariables. Si \(x_{j}\) es casi linealmente dependiente de algunos de los otros regresores, entonces \(R^{2}_{j}\) será cercano a uno y el \(VIF_{j}\) será muy alto. Generalmente, un VIF mayor de 10 indica problemas graves de multicolinealidad.
En un capítulo posterior ahondaremos más en este problema.2.7.1 Grasa corporal - factores de inflación de varianza
En el caso del modelo para grasa corporal, tenemos que los VIFs son:
## abdom adipos age
## 7.593500 7.293397 1.127968Lo que nos indica que la varianza de las estimaciones de los coeficientes se infla moderadamente debido a multicolinealidad. Esto se debe a la alta correlación entre la circunferencia del abdomen y el IMC (0.9238801).